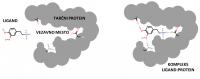
Slo-Tech - Slovenski znanstveniki se na različne načine pridružujejo mednarodnemu boju proti novemu koronavirusu. Za bralce te strani bo med zanimivejšimi gotovo računalniški pristop, v katerem lahko sodeluje vsakdo. Ekipa pod vodstvom Črtomirja Podlipnika z ljubljanske kemijske fakultete je začela projekt iskanja ligandov, ki se vežejo v različne proteinske receptorje ob okužbi z novim koronavirusom. Preverjanje, kako se ligand veže v biomolekulo, se imenuje sidranje (docking).
"Komplementarnost majhnih molekul je le prvi predpogoj, da lahko molekulo uporabimo kot zdravilo, preveriti je namreč treba tudi, kako se molekula metabolizira, kakšna je njena biološka dostopnost ter kakšna je njena toksičnost. Iskanje ustrezne molekule najlaže ponazorimo z iskanjem majhne igle v ogromni kopici sena," so pojasnili v opisu projekta. Pri tem ne pretiravajo, saj je možnih farmakoloških molekul res ogromno, po nekaterih ocenah okrog 1060. Preiskovanje čim večjega dela tega kemijskega vesolja se imenuje virtualno rešetanje. Zato se ta problem rešuje z uporabo vse dosegljive računske moči.
Ekipa je pripravila orodje, ki deluje na Windows, Mac ali Linuxu. Za delovanje potrebujemo aktivno internetno povezavo, saj paketke dobiva iz centralnega repozitorija, kamor pošilja podatke. Obdelava enega paketka traja kakšno uro. Programska oprema temelji na programu SeeSar, za katerega je licenco doniral BioSolveIt.
Omenjeni projekt pa seveda ni osamljen. V okviru projekta Folding@home, ki teče že dolgo časa, sedaj aktivno iščejo strukturo kompleksa S-pro-ACE2. Na ta način lahko predlagajo terapevtska protitelesa za virus SARS-CoV-2.
Novice » Znanost in tehnologija » Vprežite svoje računalnike v boj proti koronavirusu

vostok_1 ::
Saj komot lahko prispevate. Ampak glede na vedno več pozitivnih novic o učinkovitih zdravilih je morda bolje kam drugam vložit.
Krka bi morala sedaj talat vn na tone tega.
Krka bi morala sedaj talat vn na tone tega.
There will be chutes!
It came from the lab.
Like tears in rain. Time to die. v_1 2012-21
It came from the lab.
Like tears in rain. Time to die. v_1 2012-21
mitja_i ::
ftp://ftp.molekule.net/programs/openscience.zip
Datoteko kopirate na željeno mesto in razpakirate. Znotraj mape openscience je mapa flexx, kjer je nameščen program flexx podjetja BiosolveIt.
Program zaženete z klikom na: Run_flexx.exe
Datoteko kopirate na željeno mesto in razpakirate. Znotraj mape openscience je mapa flexx, kjer je nameščen program flexx podjetja BiosolveIt.
Program zaženete z klikom na: Run_flexx.exe
Zgodovina sprememb…
- spremenil: mitja_i ()
marvin42 ::
Potem si pa v CV napišeš "sodelovanje pri pripravi zdravila za SARS-CoV-2"
Mostly Harmless

Massacra ::

dronyx ::
Samo previdno s temi "orodji" v teh časih. Kot sem opazil se je število poskusov nateg izredno povečalo. Lahko se spomni nekdo poslat okrog orodje za "boj proti "koronavirusu", bo pa kakšen izsiljevalski virus. Pa seveda ne mislim projekt omenjen v članku.
Zgodovina sprememb…
- spremenil: dronyx ()
pirlo ::
Je neki bugasti ta program. Prvič je koristil vse threade in zračunal v par minutah. Pol ko sem dal naj avtomatsko nadaljuje, pa je cpu load na 0-1%
sivistol ::
DamijanD ::
nadlipnik ::
Za Windows je na voljo exe, ki ga poženeš in nastaviš število coreov:
ftp://ftp.molekule.net/PROGRAMS/OPENSCIENCE.zip
ftp://ftp.molekule.net/PROGRAMS/OPENSCIENCE.zip
DamijanD ::
R5 2600: flexx na 6 threadih -> obremenitev CPUja okrog 50%, en paket računa 10 - 15 minut. Pa naslednjega je tudi pravilno začel s 6timi threadi in enako obremenjenostjo CPUja.
bobby ::
Zakaj ni na GPU?
Ce eksplicitno ne odgovorim osebam PNG ali PR,..I dont care about your opinion.
pirlo ::
Je neki bugasti ta program. Prvič je koristil vse threade in zračunal v par minutah. Pol ko sem dal naj avtomatsko nadaljuje, pa je cpu load na 0-1%
Kolk threadov pa je bilo to? Kakšen CPU?
R9 3900X. Vse sem dal (24). Sem ponovno zagnal dal na 8, pa dela ok zdaj. Rabi pa okoli 7 minut.
Zgodovina sprememb…
- spremenil: pirlo ()
DamijanD ::
Glede na to, da je večina sveta tako ali drugače prizadeta zaradi tega virusa, zakaj se za take raziskave ne vpreže vseh (ali večino) super računalnikov? (Oziroma drugače, če imamo bagre, zakaj moramo plebejci kopati z žličkami)
pirlo ::
Glede na to, da je večina sveta tako ali drugače prizadeta zaradi tega virusa, zakaj se za take raziskave ne vpreže vseh (ali večino) super računalnikov? (Oziroma drugače, če imamo bagre, zakaj moramo plebejci kopati z žličkami)
Ker so verjetno ti super compi zasedeni s čim drugim. Doma pa folk lahko donira par niti in se hitro skup sešteje za par super compov.
DamijanD ::
pegasus: aha fino - saj sem samo to hotel reči, da trenutno je smiselno vse resource vržt v to
crtomirp ::
Glede na to, da je večina sveta tako ali drugače prizadeta zaradi tega virusa, zakaj se za take raziskave ne vpreže vseh (ali večino) super računalnikov? (Oziroma drugače, če imamo bagre, zakaj moramo plebejci kopati z žličkami)
Pozdravljen Damjan. V prvi vrsti gre za "Openscience for Citizen" projekt kjer sodelujejo v glavnem prostovoljci. Podoben projekt je tudi https://fold.it/ , kjer imajo
tudi vprežene uporabnike. Projekt ni strogo naravosloven, ampak vsebuje tudi sociološko komponento. Seveda si želimo, da bi našli ustrezno majhno molekulo, ki bi zavrla delovanje koronavirusa, če pri tem sodelujejo prostovoljci, ki vsekakor niso plebejci, je toliko lepše. Seveda bo čas tudi, da se superačunalniki vključijo v projekt
in jih bomo tudi vključili. Mogoče bomo poganjali eno tarčo kot openscience drugo pa s superačunalniki. Vendar potrebujemo res ogromno zbirko spojin zaenkrat jih je
v naši knjižnici okoli 310 000. Škoda, da program za sidranje ne uporablja GPU-jev, če bi jih bi se mogoče vključil tudi kakšen kripto-rudar in namesto kripto-valut
dan ali dva rudaril spojine.
Glede pomankljivosti, ki jih imajo naši programčki pa je zadeva takšna. Začetnega razvoj skript sem se lotil sam kot non-IT strokovnjak, pred šestimi dnevi,
pravtako je bilo potrebno pripraviti tudi repozitorij , ker sem zaposlen na Fakulteti za kemijo imam sedaj tudi mnogo drugih obveznosti, ..... Sedaj sta razvoj skript prevzela kolega, ki sta bolj podučena v IT zadevah kot jaz osebno in dnevno iščeta nove in nove rešitve. Tako, da so seveda možne tudi kakšne pomankljivosti,
idealno bi bilo, da bi program potekal v ozadju, sicer bi za obdelavo paketka potrebovali nekaj več časa ampak bi lahko uporabnik lahko računalnik uporabljal bolj nemoteno tudi za druge operacije. Seveda bi bilo dobro vsaj za Win64 in MacOS narediti grafični klient, kjer bi bila prikazana statistika, bile bi možne razne nastavitve ipd.
Lep pozdrav,
Črtomir Podlipnik, predstavnik projekta
Zgodovina sprememb…
- spremenilo: crtomirp ()
DamijanD ::
Če sem pravilno razumel je cilj s tem pregledati 310 000 spojin. Trenutno smo pa na 1212 (če prav sklepam iz programčka). A je kje tudi kakšna statistika, koliko jih sodeluje ipd?
crtomirp ::
Pozdravljan Damjan 1212 je številka paketka, ki je obdelan v paketku je 250 spojin.
Danes gre hitreje kot sem mislil, da bo šlo grem pripravljat nove paketke.
Danes gre hitreje kot sem mislil, da bo šlo grem pripravljat nove paketke.
Zgodovina sprememb…
- spremenilo: crtomirp ()
Tuhi ::
DamijanD ::
Sem videl potem, da niso zaporedne številke. Na enem računalniku sem zdajle dobil:
3D_structures_1337.sdf:31: ERROR: Molecule Dibutyryl-cAMP.cdx: Element error encountered
3D_structures_1337.sdf:31: ERROR: Entry 32 (Dibutyryl-cAMP.cdx) contained errors
3D_structures_1337.sdf:32: ERROR: Molecule 8-Bromo-cAMP.cdx: Element error encountered
3D_structures_1337.sdf:32: ERROR: Entry 33 (8-Bromo-cAMP.cdx) contained errors
3D_structures_1337.sdf:68: ERROR: Molecule : Element error encountered
3D_structures_1337.sdf:68: ERROR: Entry 69 contained errors
3D_structures_1337.sdf:102: ERROR: Molecule S3770 Sodium Aescinate.cdx: Element error encountered
3D_structures_1337.sdf:102: ERROR: Entry 103 (S3770 Sodium Aescinate.cdx) contained errors
3D_structures_1337.sdf:115: ERROR: Molecule : Element error encountered
3D_structures_1337.sdf:115: ERROR: Entry 116 contained errors
3D_structures_1337.sdf:229: ERROR: Molecule s4075.cdx: Element error encountered
3D_structures_1337.sdf:229: ERROR: Entry 230 (s4075.cdx) contained errors
3D_structures_1337.sdf:31: ERROR: Molecule Dibutyryl-cAMP.cdx: Element error encountered
3D_structures_1337.sdf:31: ERROR: Entry 32 (Dibutyryl-cAMP.cdx) contained errors
3D_structures_1337.sdf:32: ERROR: Molecule 8-Bromo-cAMP.cdx: Element error encountered
3D_structures_1337.sdf:32: ERROR: Entry 33 (8-Bromo-cAMP.cdx) contained errors
3D_structures_1337.sdf:68: ERROR: Molecule : Element error encountered
3D_structures_1337.sdf:68: ERROR: Entry 69 contained errors
3D_structures_1337.sdf:102: ERROR: Molecule S3770 Sodium Aescinate.cdx: Element error encountered
3D_structures_1337.sdf:102: ERROR: Entry 103 (S3770 Sodium Aescinate.cdx) contained errors
3D_structures_1337.sdf:115: ERROR: Molecule : Element error encountered
3D_structures_1337.sdf:115: ERROR: Entry 116 contained errors
3D_structures_1337.sdf:229: ERROR: Molecule s4075.cdx: Element error encountered
3D_structures_1337.sdf:229: ERROR: Entry 230 (s4075.cdx) contained errors
hlod_the_one ::
Pozdravljan Damjan 1212 je številka paketka, ki je obdelan v paketku je 250 spojin.
Danes gre hitreje kot sem mislil, da bo šlo grem pripravljat nove paketke.
Kako bi naj to orodje pognal na linuxu?
Prenesi si ftp://ftp.molekule.net/PROGRAMS/FlexX_linux64bit.zip
Extract-aj datoteke
Odpri terminal in pojdi v `flexx` mapo
Zaženi z ukazom `./run_flexx.sh`
Pri meni je bila težava če sem pognal "update" na Linux (Ubuntu 18.04.4 LTS) in Windows 10.
LP.
crtomirp ::
Bom moral pognati še en filter za te spojine da jih prečistim oz
da ven zmečem vse ligande ki so čudni ...
Bom videl če moj laptop to zmore.
da ven zmečem vse ligande ki so čudni ...
Bom videl če moj laptop to zmore.
2g00d4u ::
A je mogoče da smo oddelali vse pakete, ker mi neprestano prenaša pakete z napakami?
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_229.sdf -o OUT_229.sdf -v 4 --max-nof-conf 1 --thread-count 24
Loading protein
Loading reference ligand.
ERROR: No entries loaded from file TEST_REF.sdf
ERROR: No reference ligand available.
Error flexing package (stdout) 229 : exit status 1
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_229.sdf -o OUT_229.sdf -v 4 --max-nof-conf 1 --thread-count 24
Loading protein
Loading reference ligand.
ERROR: No entries loaded from file TEST_REF.sdf
ERROR: No reference ligand available.
Error flexing package (stdout) 229 : exit status 1
Zgodovina sprememb…
- spremenil: 2g00d4u ()
aky ::
Ja, en kup errojev, potem pa spet pride paket, ki je za narediti...
...
Prerequired files downloaded
Calculating package 367
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_367.sdf -o OUT_367.sdf -v 4 --max-nof-conf 1 --thread-count 48
Loading protein
Loading reference ligand.
ERROR: No entries loaded from file TEST_REF.sdf
ERROR: No reference ligand available.
Error flexing package (stdout) 367 : exit status 1
Prerequired files downloaded
Calculating package 379
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_379.sdf -o OUT_379.sdf -v 4 --max-nof-conf 1 --thread-count 48
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
StratOS ::
Prerequired files downloaded
Calculating package 372
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_372.sdf -o OUT_372.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
ERROR: No entries loaded from file TEST_REF.sdf
ERROR: No reference ligand available.
Error flexing package (stdout) 372 : exit status 1
Prerequired files downloaded
Calculating package 383
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_383.sdf -o OUT_383.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
Calculating package 372
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_372.sdf -o OUT_372.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
ERROR: No entries loaded from file TEST_REF.sdf
ERROR: No reference ligand available.
Error flexing package (stdout) 372 : exit status 1
Prerequired files downloaded
Calculating package 383
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_383.sdf -o OUT_383.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
"Multitasking - ability to f##k up several things at once."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
Tuhi ::
hlod_the_one je izjavil:
Pozdravljan Damjan 1212 je številka paketka, ki je obdelan v paketku je 250 spojin.
Danes gre hitreje kot sem mislil, da bo šlo grem pripravljat nove paketke.
Kako bi naj to orodje pognal na linuxu?
Prenesi si ftp://ftp.molekule.net/PROGRAMS/FlexX_linux64bit.zip
Extract-aj datoteke
Odpri terminal in pojdi v `flexx` mapo
Zaženi z ukazom `./run_flexx.sh`
Pri meni je bila težava če sem pognal "update" na Linux (Ubuntu 18.04.4 LTS) in Windows 10.
LP.
Dobim tole:
/ftp.molekule.net/PROGRAMS$ ./run_flexx.sh
Welcome to the CITIZEN SCIENCE COVID-19 v0.4.1
lib/run_flexx: line 40: curl: command not found
lib/run_flexx: line 41: [: 0.4.1: unary operator expected
lp
HotBurek ::
Evo, jst se isto lotevam tega in imam isti problem.
Prvo bi zgolj predlagal, da dodajte, da če ni nameščenega curl, naj program naredi failover na wget.
Jst sem pri namestitvi curl naletel na neke dependency izzive, kjer mi želi odstranit ModgoDB server...
Skratka, če imaš možnost, namesti curl (apt-get install curl).
Tisti, ki pa razvijate ta program; if curl is missing, try with wget.
Prvo bi zgolj predlagal, da dodajte, da če ni nameščenega curl, naj program naredi failover na wget.
Jst sem pri namestitvi curl naletel na neke dependency izzive, kjer mi želi odstranit ModgoDB server...
Skratka, če imaš možnost, namesti curl (apt-get install curl).
Tisti, ki pa razvijate ta program; if curl is missing, try with wget.
root@debian:/# iptraf-ng
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window
Zgodovina sprememb…
- spremenilo: HotBurek ()
Tuhi ::
Evo, jst se isto lotevam tega in imam isti problem.
Prvo bi zgolj predlagal, da dodajte, da če ni nameščenega curl, naj program naredi failover na wget.
Jst sem pri namestitvi curl naletel na neke dependency izzive, kjer mi želi odstranit ModgoDB server...
Skratka, če imaš možnost, namesti curl (apt-get install curl).
Tisti, ki pa razvijate ta program; if curl is missing, try with wget.
Po inštalaciji curl-a sedaj dela..
tnx
HotBurek ::
Jst tudi rešil.
Mi je pa sicer pri zagonu javilo tole:
environment: line 94: [: too many arguments
environment: line 108: [: too many arguments
A dela normalno.
Imam za ustvarjalce vprašanje;
Would you like to keep the FlexX output files? ([Y]es/[n]o) Y
Za kakšne output fajle gre tu?
Pa idealno bi bilo, da bi prikazovalo nekakšen procen od nečesa, s kakšno hitrostjo rešuje (da lahko malo primerjamo hitrosti), kje in kaj program dela. Vsaj pri meni trenutno še ni nobenih premikajočih številk, da bi z lahkoto videl, da program živi, dela in se premika naprej. Mogoče je zaciklan v while(true) zaniki. :)
htop sicer lepo pokaže cpu na 100%
Pa za editor: če lahko na konvu novice dodate link do ZIP fajla; ene za winblows, drugi za linukse.
ftp://ftp.molekule.net/PROGRAMS/OPENSCIENCE.zip
ftp://ftp.molekule.net/PROGRAMS/FlexX_linux64bit.zip
Mi je pa sicer pri zagonu javilo tole:
environment: line 94: [: too many arguments
environment: line 108: [: too many arguments
A dela normalno.
Imam za ustvarjalce vprašanje;
Would you like to keep the FlexX output files? ([Y]es/[n]o) Y
Za kakšne output fajle gre tu?
Pa idealno bi bilo, da bi prikazovalo nekakšen procen od nečesa, s kakšno hitrostjo rešuje (da lahko malo primerjamo hitrosti), kje in kaj program dela. Vsaj pri meni trenutno še ni nobenih premikajočih številk, da bi z lahkoto videl, da program živi, dela in se premika naprej. Mogoče je zaciklan v while(true) zaniki. :)
htop sicer lepo pokaže cpu na 100%
Pa za editor: če lahko na konvu novice dodate link do ZIP fajla; ene za winblows, drugi za linukse.
ftp://ftp.molekule.net/PROGRAMS/OPENSCIENCE.zip
ftp://ftp.molekule.net/PROGRAMS/FlexX_linux64bit.zip
root@debian:/# iptraf-ng
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window
Zgodovina sprememb…
- spremenilo: HotBurek ()
poweroff ::
Pozdravljan Damjan 1212 je številka paketka, ki je obdelan v paketku je 250 spojin.
Danes gre hitreje kot sem mislil, da bo šlo grem pripravljat nove paketke.
Kako bi naj to orodje pognal na linuxu?
./run_flexx.sh
Welcome to the CITIZEN SCIENCE COVID-19 v0.4.1
Your current machine has 8 available threads
Please enter how many threads you would like this software to use (1-8/[A]ll) 4
Would you like to keep the FlexX output files? ([Y]es/[n]o)
Please answer yes or no.
Would you like to keep the FlexX output files? ([Y]es/[n]o) yes
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 3 0 3 0 0 8 0 --:--:-- --:--:-- --:--:-- 8
environment: vrstica 94: [: preveč argumentov
environment: vrstica 108: [: preveč argumentov
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
<html>
<head><title>413 Request Entity Too Large</title></head>
<body bgcolor="white">
<center><h1>413 Request Entity Too Large</h1></center>
<hr><center>nginx/1.14.0 (Ubuntu)</center>
</body>
</html>
...
...
Would you like to calculate the next package? (Y/n) % Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 3 0 3 0 0 12 0 --:--:-- --:--:-- --:--:-- 12
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
<html>
<head><title>413 Request Entity Too Large</title></head>
<body bgcolor="white">
<center><h1>413 Request Entity Too Large</h1></center>
<hr><center>nginx/1.14.0 (Ubuntu)</center>
</body>
</html>
sudo poweroff
2g00d4u ::
Koliko nas sploh računa te zadeve, ker imam občutek da nas več enake pakete računa. Vsakič ko pride do cca 1300 paketa gre od začetka. Torej ne povrsti ampak vedno druge pakete. Trenutno imam preko 100 output paketov in dvomim da nas je tako malo.
APPluk ::
Tole bi lahko mleli zdaj vsi računalniki po fakultetah pa srednjih šolah 
Meni je tudi cudno, ker sem začel pri paketu čez 1000, zdaj pa gre spet od začetka po okrog +60 glede na prejšnjo št. paketka.
Meni je tudi cudno, ker sem začel pri paketu čez 1000, zdaj pa gre spet od začetka po okrog +60 glede na prejšnjo št. paketka.
bedarija ::
eh kompi na faksih tečejo na pentiumu, je tvoj mobitel močnejši =) kakorkoli je to fajn zadeva, ravno zdej vsi čepijo doam in uporabljajo kompe tak da gre to samo ponoči.
StratOS ::
Welcome to the CITIZEN SCIENCE COVID-19 0.3
to start press enter
Using latest version
Your computer is capable of using 4 simultaneous threads, how many of them do you want to utilize?
4
Prerequired files downloaded
Calculating package 865
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_865.sdf -o OUT_865.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
2020/03/20 19:15:46 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
¸¸¸¸Y
Prerequired files downloaded
Calculating package 402
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_402.sdf -o OUT_402.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
WARNING: Skipped 1 molecules without 3D coordinates.
2020/03/20 20:13:52 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
WARNING: Skipped 1 molecules without 3D coordinates.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 1212
Zakaj ne teče naprej tako kot bi moralo ?
Še eno par napak :
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 1212
2020/03/20 20:19:16 Error storing package file, aborting net/http: request canceled (Client.Timeout exceeded while reading body)
Prerequired files downloaded
Calculating package 1219
2020/03/20 20:20:05 Error storing package file, aborting net/http: request canceled (Client.Timeout exceeded while reading body)
Prerequired files downloaded
Calculating package 1223
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_1223.sdf -o OUT_1223.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
to start press enter
Using latest version
Your computer is capable of using 4 simultaneous threads, how many of them do you want to utilize?
4
Prerequired files downloaded
Calculating package 865
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_865.sdf -o OUT_865.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
2020/03/20 19:15:46 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
¸¸¸¸Y
Prerequired files downloaded
Calculating package 402
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_402.sdf -o OUT_402.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
WARNING: Skipped 1 molecules without 3D coordinates.
2020/03/20 20:13:52 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
WARNING: Skipped 1 molecules without 3D coordinates.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 1212
Zakaj ne teče naprej tako kot bi moralo ?
Še eno par napak :
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 1212
2020/03/20 20:19:16 Error storing package file, aborting net/http: request canceled (Client.Timeout exceeded while reading body)
Prerequired files downloaded
Calculating package 1219
2020/03/20 20:20:05 Error storing package file, aborting net/http: request canceled (Client.Timeout exceeded while reading body)
Prerequired files downloaded
Calculating package 1223
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_1223.sdf -o OUT_1223.sdf -v 4 --max-nof-conf 1 --thread-count 4
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
"Multitasking - ability to f##k up several things at once."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
Zgodovina sprememb…
- spremenila: StratOS ()
HotBurek ::
Vidim, da nekaterim izpisuje razne "file upload progress" korake. Jst tega kr nimam.
Da pa avtomatsko obdeluje naslednji paket; jst sem dal Y samo prvikrat, ko vpraša, za ostale gre pa po par sekundah avtomatsko naprej.
Da pa avtomatsko obdeluje naslednji paket; jst sem dal Y samo prvikrat, ko vpraša, za ostale gre pa po par sekundah avtomatsko naprej.
root@debian:/# iptraf-ng
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window
fatal: This program requires a screen size of at least 80 columns by 24 lines
Please resize your window

Evolve ::
ste blesavi...
tole je bait za majnanje bitkojnov
tole je bait za majnanje bitkojnov
Zgodovina sprememb…
- predlagalo izbris: jest10 ()
StratOS ::
Prerequired files downloaded
Calculating package 3150
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_3150.sdf -o OUT_3150.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
3D_structures_3150.sdf:90: WARNING: Taking largest component (39 atoms)
3D_structures_3150.sdf:92: WARNING: Taking largest component (37 atoms)
3D_structures_3150.sdf:242: WARNING: Taking largest component (41 atoms)
3D_structures_3150.sdf:243: WARNING: Taking largest component (47 atoms)
3D_structures_3150.sdf:244: WARNING: Taking largest component (36 atoms)
3D_structures_3150.sdf:245: WARNING: Taking largest component (33 atoms)
3D_structures_3150.sdf:246: WARNING: Taking largest component (38 atoms)
3D_structures_3150.sdf:247: WARNING: Taking largest component (29 atoms)
WARNING: Skipped 192 molecules without 3D coordinates.
2020/03/21 06:49:38 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
3D_structures_3150.sdf:90: WARNING: Taking largest component (39 atoms)
3D_structures_3150.sdf:92: WARNING: Taking largest component (37 atoms)
3D_structures_3150.sdf:242: WARNING: Taking largest component (41 atoms)
3D_structures_3150.sdf:243: WARNING: Taking largest component (47 atoms)
3D_structures_3150.sdf:244: WARNING: Taking largest component (36 atoms)
3D_structures_3150.sdf:245: WARNING: Taking largest component (33 atoms)
3D_structures_3150.sdf:246: WARNING: Taking largest component (38 atoms)
3D_structures_3150.sdf:247: WARNING: Taking largest component (29 atoms)
WARNING: Skipped 192 molecules without 3D coordinates.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 3263
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_3263.sdf -o OUT_3263.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
Calculating package 3150
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_3150.sdf -o OUT_3150.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
3D_structures_3150.sdf:90: WARNING: Taking largest component (39 atoms)
3D_structures_3150.sdf:92: WARNING: Taking largest component (37 atoms)
3D_structures_3150.sdf:242: WARNING: Taking largest component (41 atoms)
3D_structures_3150.sdf:243: WARNING: Taking largest component (47 atoms)
3D_structures_3150.sdf:244: WARNING: Taking largest component (36 atoms)
3D_structures_3150.sdf:245: WARNING: Taking largest component (33 atoms)
3D_structures_3150.sdf:246: WARNING: Taking largest component (38 atoms)
3D_structures_3150.sdf:247: WARNING: Taking largest component (29 atoms)
WARNING: Skipped 192 molecules without 3D coordinates.
2020/03/21 06:49:38 Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
3D_structures_3150.sdf:90: WARNING: Taking largest component (39 atoms)
3D_structures_3150.sdf:92: WARNING: Taking largest component (37 atoms)
3D_structures_3150.sdf:242: WARNING: Taking largest component (41 atoms)
3D_structures_3150.sdf:243: WARNING: Taking largest component (47 atoms)
3D_structures_3150.sdf:244: WARNING: Taking largest component (36 atoms)
3D_structures_3150.sdf:245: WARNING: Taking largest component (33 atoms)
3D_structures_3150.sdf:246: WARNING: Taking largest component (38 atoms)
3D_structures_3150.sdf:247: WARNING: Taking largest component (29 atoms)
WARNING: Skipped 192 molecules without 3D coordinates.
Done calculating page
File uploaded page
Do you want to continue automatically next time? (y/n)
y
Prerequired files downloaded
Calculating package 3263
lib\FLEXX\flexx -p TEST_PRO.pdb -r TEST_REF.sdf -i 3D_structures_3263.sdf -o OUT_3263.sdf -v 4 --max-nof-conf 1 --thread-count 2
Loading protein
Loading reference ligand.
Preparing protein and determine active site.
Run docking.
"Multitasking - ability to f##k up several things at once."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
"It works better if you plug it in."
"The one who is digging the hole for the other to fall in is allready in it."
Vredno ogleda ...
| Tema | Ogledi | Zadnje sporočilo | |
|---|---|---|---|
| Tema | Ogledi | Zadnje sporočilo | |
| » | Folding@Home (strani: 1 2 )Oddelek: Znanost in tehnologija | 11829 (4435) | quadrok3100m |
| » | Kitajska nad koronavirus tudi z dezinfekcijo in uničevanjem bankovcevOddelek: Novice / Znanost in tehnologija | 6027 (4026) | Cash |
| » | Ustvarili bakterijo s tremi različnimi baznimi pari v DNKOddelek: Novice / Znanost in tehnologija | 10617 (9097) | tikitoki |
| » | Živčni strupi v Siriji: biokemijsko ozadjeOddelek: Novice / Znanost in tehnologija | 19353 (15680) | Afo |
| » | Iskanje novih zdravil s simulacijami in evolucijoOddelek: Znanost in tehnologija | 4281 (3583) | dukenukem |