Slo-Tech - Da Nobelove nagrade naposled prepoznavajo pomen računalniških orodij v znanosti, priča tudi letošnja nagrada za kemijo, ki jo bodo prejeli David Baker z Univerze Washington v Seattlu ter Demis Hassabis in John M. Jumper iz podjetja DeepMind. Baker je prejel polovico nagrade za račualniško načrtovanje proteinov, Hassabis in Jumper pa za napovedi strukture proteinov.
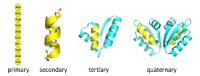
Proteini so osnovni gradniki živih bitij, ki jim omogočajo najrazličnejše strukturne prilagoditve. Lasje, nohti, koža in številna druga tkiva imajo svoje lastnosti zaradi proteinov, ki jih gradijo. Izjemna raznolikost je pravzaprav neverjetna, saj vse proteine gradi zgolj 20 različnih aminokislin. Vrstni red, v katerem se te aminokisline povezujejo v proteine, je zapisan v DNK. Aminokisline se nato povežejo s peptidnimi vezmi, a čarovnija se zgodi šele za tem. Zaporedje aminokislin imenujemo primarna struktura in ni posebej razburljiva, nato pa se nastali polimer zvije v prostoru, s čimer pridobi sekundarno in terciarno (lahko pa tudi kvartarno, če gre za velike proteine) strukturo. Proteini potem segajo od čvrstih las do encimov v celicah, ki pomagajo pri presnovi glukoze.
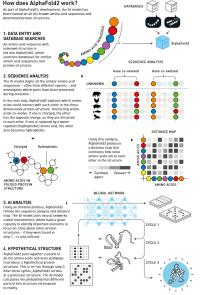
Iz zapisa DNK je trivialno ugotoviti, katere aminokisline bodo v tvorile protein. Obstajajo tabele kodonov, ki to natančno opišejo. Raznolikost je res izjemna: iz 20 aminokislin poznamo že 200 milijonov različnih proteinov. A bistveno težja zagonetka je napoved, kako se bo aminokislinsko zaporedje zvilo v prostoru - napoved tridimenzionalne strukture je bila dolgo vrsto let nemogoča, zato so se morali raziskovalci naslanjati na eksperimentalne tehnike (npr. rentgensko kristalografijo). A meritve so pogosto nemogoče, če proteina ni možno izolirati. Druga vprašanje je seveda ciljani dizajn, torej iskanje zaporedja aminokislin, ki bi dalo protein s točno določeno funkcijo. Recimo encim, ki bi razgrajeval nafto v madežih po razlitjih na morju.
Proteini se vedno zvijejo enako. Četudi ga razvijemo, bo nato zavzel enako terciarno strukturo, je že leta 1961 odkril Anfinsen. To je sicer dobro, ker daje robustnost življenju. Hkrati to pomeni, da je tridimenzionalna struktura nekako zapisana v zaporedju aminokislin in bi morala biti v teoriji izračunljiva. Možnosti je ogromno in Anfinsen je ocenil, da je načinov, kako se protein zvije, več od števila atomov v vesolju, a bo vedno našel isti način. Ga lahko tudi mi? Dolgo časa je kazalo, da ne, nato pa so na pomoč priskočili računalniki. Od leta 1994 poteka tekmovanje CASP (Critical Assessment of Protein Structure Prediction), na katerem se pomerijo različni računalniški programi za napoved strukture, ki se trudijo na istih zaporedji aminokislin, za katere so strukturo proteina pred tem eksperimentalno določini, a podatek ni javno znan. Dolgo so bili rezultati klavrni, nato pa je leta 2018 na tekmovanje vstopil Demis Hassabis s podjetjem DeepMind, ki ga sicer poznamo po strojnem učenju in programih za igranje šaha, goja in drugih iger. DeepMind je resda premagal človeškega svetovnega prvaka v goju, a to ni bil cilj podjetja. Njihov program AlphaFold je namesto tega začel preiskovati zvijanje proteinov - in pri tem briljiral.
Ključni prispevek je dodal John Jumper, ki se je pridružil podjetju leta 2017. Sodelova je pri razvoju AlphaFolda 2, ki je uporabljal tehnologijo transformerjev. Nevronske mreže in umetna inteligenca so razvozlali uganko, kako se bodo proteini zvili. AlphaFold 2 je leta 2020 že dosegel rezultate, ki so bili primerljivi z eksperimentalnimi meritvami. Zvijanje proteinov je danes zadovoljivo rešen problem. Poznavanje zvijanja proteinov pa omogoča, da problem obrnemo. Ta ideja se je Davidu Bakerju porodila že davnega leta 1998, še preden so obstajali transformerji in AlphaFold, ko se je trudil s svojim programom Rosetta, ki je bil za tisti čas precej dober. Medtem ko so drugi raziskovalci modificirali obstoječe proteine, da bi izboljšali njihove funkcije, je Bakerjeva skupina želela poizkusiti od začetka. Ali bi lahko dizajnirali povsem nov protein, ki bi počel kaj koristnega? Področje se imenuje de novo dizajn proteinov. Eden prvih rezultatov je bil novi protein Top7, ki ga sestavlja 93 aminokislin in o katerem so prvikrat poročali leta 2003. Kasneje so dizajnirali še več proteinov, kodo za Rosetto pa so javno objavili. Enako velja za AlphaFold 2.
Orodja obstajajo. Določanje strukture proteinov, ki je včasih terjalo več let, je sedaj izvedljivo v nekaj minutah. Dizajn novih proteinov sedaj postaja čedalje bolj uveljavljena tehnika, ki omogoča najrazličnejše uporabe: selektivno zaznavanje fentanila, izdelava imitacij virusov za cepljenje, načrtovanje biosenzorjev, molekulskih motorjev itd. In morda si bomo v prihodnosti lahko dizajnirali in sintetizirali proteine, ki bodo povsem a la carte po funkciji.
Novice » Znanost in tehnologija » Nobelova nagrada za kemijo 2024
.:joco:. ::
Kako nam poznavanje 3d strukture proteina pomaga ugotovit kaj naj bi protein delal? Še vedno je treba probat? Še vedno ne veš kaj vse bo zraven delal, česar nisi predvideval?
"Is science true?"
You don't get it.
Science is the process of trying to find out what's true.
You don't get it.
Science is the process of trying to find out what's true.
tikitoki ::
Kako nam poznavanje 3d strukture proteina pomaga ugotovit kaj naj bi protein delal? Še vedno je treba probat? Še vedno ne veš kaj vse bo zraven delal, česar nisi predvideval?
Primerjas s strukturo proteinov z znano funkcijo.
Metoda ima pomankljivosti, vendar ima tudi prednosti. Iz zaporedja aminokislin lahko napoves vlogo proteina. Seveda moras napoved preveriti. Imas pa neko izhodisce, kar olajsa zadevo.
Imas tudi druge pristope, ki se uporabljajo za napovedovanje interakcij. Kjer moras rezultate seveda ponovno preveriti. Vendar dobis izhodisce kaj bos preveril in vitro, al pa in vivo.
Zgodovina sprememb…
- spremenilo: tikitoki ()
pegasus ::
DeepMind je objavil predavanja iz njihovega dogodka "AI for Science". Fascinantni pogovori:
https://www.youtube.com/@Google_DeepMin...
https://www.youtube.com/@Google_DeepMin...
Vredno ogleda ...
| Tema | Ogledi | Zadnje sporočilo | |
|---|---|---|---|
| Tema | Ogledi | Zadnje sporočilo | |
| » | Google DeepMind predstavil algoritem AlphaFold 3Oddelek: Novice / Znanost in tehnologija | 4129 (1448) | pegasus |
| » | Nobelova nagrada za kemijo 2024Oddelek: Novice / Znanost in tehnologija | 4087 (2349) | pegasus |
| » | AlphaFold izračunal strukturo vseh proteinovOddelek: Novice / Znanost in tehnologija | 7208 (4656) | pegasus |
| » | Alphabet ustanovil podjetje za raziskave zdravilOddelek: Novice / Znanost in tehnologija | 4501 (3671) | Prospekt |
| » | DeepMindov algoritem MuZero se uči kot otrokOddelek: Novice / Znanost in tehnologija | 13547 (10645) | 7982884e |